La lattato deidrogenasi o LDH (EC 1.1.1.27) è una famiglia di ossidoreduttasi che catalizza la conversione reversibile del piruvato a lattato, con la concomitante interconversione di NADH e NAD+, i quali agiscono da cofattori.
Sono enzimi a struttura tetramerica, dove ogni subunità è dotata di attività catalitica. Le subunità, codificate da geni distinti, si possono combinare in proporzioni differenti a dare isoenzimi con proprietà cinetiche e regolatorie specifiche.[1]
La lattato deidrogenasi è presente in quasi tutti i tessuti animali e nelle piante, ma anche nei microorganismi. Sebbene sia presente per la maggior parte nel citosol, la sua presenza è stata dimostrata anche nei mitocondri, dove catalizza l’ossidazione del lattato a piruvato, e nei perossisomi.[2][3]
Nell’uomo in tessuti differenti prevalgono isoenzimi differenti, sulla base dello specifico fenotipo metabolico del tessuto.
La lattato deidrogenasi è un enzima cruciale nel metabolismo cellulare, essendo coinvolta nella produzione di energia dai carboidrati in condizioni anaerobiche, nella sintesi del glucosio dal lattato e nell’utilizzo dello scheletro carbonioso del lattato a fini energetici in condizioni aerobiche.[1][4]
Indice
Geni
Nei mammiferi esistono diversi geni che codificano per le subunità della lattato deidrogenasi, indicati come LDHA, LDHB, LDHC, LDHx e LDHD.
I primi quattro codificano enzimi che riconoscono come substrato gli L-isomeri dell’acido lattico, la principale forma enantiomerica della molecola presente nei vertebrati, e hanno come cofattore il NAD, mentre LDHD codifica per un enzima che riconosce come substrato l’isomero D dell’acido lattico e ha come cofattore il FAD.[5][6]
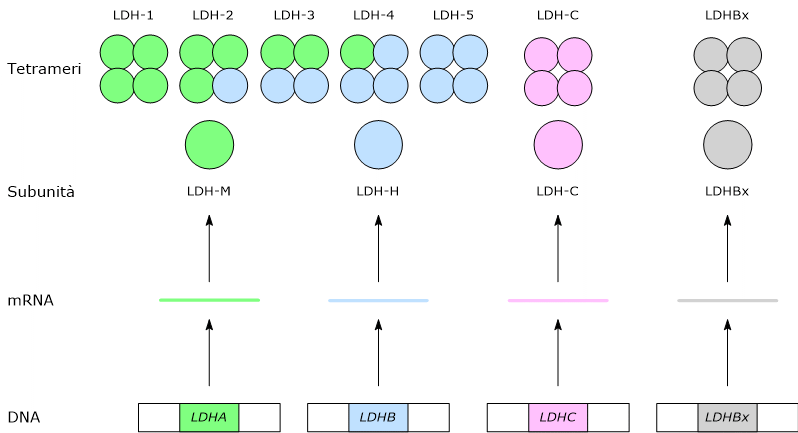
LDHA, presente sul cromosoma 11p15.4, codifica per subunità M, dall’inglese muscle, mentre LDHB, presente sul cromosoma 12p12.2-p12.1, codifica per la subunità H, dall’inglese heart.[2][7]
LDHC, presente sul cromosoma 11p15.5-p15.3 e probabilmente derivante dalla duplicazione del gene LDHA, codifica per una subunità indicata come C.[8]
Infine, LDHx codifica per la subunità specifica dei perossisomi.[2] LDHx è la forma readthrough del gene LDHB; infatti quello che succede è che il ribosoma, nel momento in cui arriva al codone di stop dell’mRNA per la subunità H inserisce un aminoacido anziché terminare la traduzione, che procede aggiungendo altri sette aminoacidi che costituiscono il targeting perossisomiale di quella che sarebbe una normale subunità H.[3]
Struttura
Gli enzimi appartenenti alla famiglia della lattato deidrogenasi hanno struttura oligomerica, nello specifico sono tetrameri derivanti dall’assemblaggio di subunità, di circa 35 kD, che possono essere di uno stesso tipo o due tipi differenti. Ognuna subunità è dotata di attività catalitica, quindi il tetramero presenta quattro siti attivi. Tuttavia le subunità, prese singolarmente, risultano cataliticamente inattive.[7][9]
Considerando le subunità H e M, le loro strutture primarie sono per circa il 75% identiche, e di conseguenza anche la loro struttura tridimensionale risulta molto simile, ma con piccole differenze a livello del sito di legame per il substrato che portano a grosse differenze nelle proprietà cinetiche delle proteine.[1][10] Altra conseguenza delle differenze nella struttura primaria riguarda la carica netta di superficie che risulta essere di -6 per la subunità M e +1 per la H.[11]
La struttura secondaria è costituita per circa un 40% da alfa-eliche e un 23% da foglietti beta, a formare strutture beta-alfa-beta, con i foglietti beta paralleli come componente principale.[12]
Nell’uomo gli isoenzimi prevalenti sono quelli formati dalle subunità H e M. L’assemblaggio, in proporzioni differenti, delle due subunità porta alla formazione di cinque isoenzimi, indicati come:
- H4 o LDH-1;
- H3M1 o LDH-2;
- H2M2 o LDH-3;
- H1M3 o LDH-4;
- M4 o LDH-5.[4]
 La subunità C va a formare una sesta isoforma, omotetramerica, indicata come LDH-6 o C4.[8]
La subunità C va a formare una sesta isoforma, omotetramerica, indicata come LDH-6 o C4.[8]
Una settima isoforma deriva dall’assemblaggio della subunità LDHx.[3][8]
Reazione
La lattato deidrogenasi catalizza l’interconversione del piruvato e lattato, che le forme ionizzate rispettivamente dell’acido piruvico e dell’acido lattico, e del NADH e NAD+. In entrambe i casi si verifica la rimozione, da quello che è l’agente riducente, di due atomi di idrogeno, e il successivo trasferimento di uno ione idruro, ossia un protone e due elettroni, all’agente ossidante, mentre il protone rimanente viene rilasciato nel mezzo acquoso. L’enzima è in grado di aumentare la velocità di reazione di 14 volte.[5]
Quando la reazione procede da piruvato a lattato, il primo passaggio è il legame del NADH, l’agente riducente, all’enzima, legame che è seguito da cambiamenti conformazionali che facilitano la formazione di legami idrogeno tra specifici residui delimitanti il sito attivo e il carbonile del piruvato, ossia il C2, e la successiva interazione tra NADH e il piruvato.[13] A questo punto si verifica il trasferimento di uno ione idruro dall’anello nicotinamidico del NADH al C2 del piruvato, che viene quindi ridotto a lattato. Ciò provoca l’ossidazione del coenzima e la neutralizzazione della carica positiva dell’azoto dell’anello nicotinamidico, e la riduzione del piruvato a lattato. Pertanto, in questo caso il C2 del piruvato viene ridotto da chetone ad alcol.
Quando la reazione procede in direzione opposta lo ione idruro viene trasferito dal C2 del lattato, che in questo caso è l’agente riducente, al C-4 dell’anello nicotinamidico. In questo caso il C2 del lattato è ossidato da alcool a chetone.[14][15]
Sito attivo
Il sito attivo delle subunità H e M ha una struttura ben conservata nelle differenti specie, con gli stessi aminoacidi che partecipano alla reazione.[16] Tra questi, non solo per la lattato deidrogenasi umana, ma anche per quella di molte altre specie, c’è His-193, aminoacido che si trova vicino al sito di legame per il coenzima.
Tuttavia, le piccole differenze nella struttura primaria delle due subunità, determinano differenze nelle proprietà biochimiche. Tra le differenze, una in particolare risulta cruciale per le proprietà catalitiche: una alanina della subunità M è sostituita, nella subunità H, da una glutammina, ossia un aminoacido non polare è sostituito da uno polare.[11] Di seguito una breve rassegna delle differenza catalitiche tra le due subunità.
La subunità H lega il substrato più velocemente della subunità M, ma ha, rispetto alla M un’attività catalitica circa cinque volte inferiore.
La subunità M ha una maggiore affinità per il piruvato, favorendo quindi la sintesi di lattato e NAD+, e non è inibita da alte concentrazioni di piruvato. Di contro, la subunità H ha un’affinità maggiore per il lattato, il che favorisce la sintesi di piruvato e NADH, mentre è inibita da elevate concentrazioni di piruvato.[17][18]
Da tutto ciò consegue che le proprietà cinetiche/catalitiche dei differenti isoenzimi dipendono dalla prevalenza di una delle due subunità.[19]
Regolazione ad opera del piruvato e lattato
Gli isoenzimi della lattato deidrogenasi sono soggetti a inibizione da parte del piruvato e lattato.
Gli isoenzimi dove predomina la subunità H sono inibiti da concentrazioni di piruvato più basse rispetto a quelli dove predomina la subunità M. Ad esempio, H4 è inibito da concentrazioni di piruvato pari a circa 0,2 mM, mentre M4 è debolmente inibito dal piruvato fino a concentrazioni di 5 mM.
Di contro, H4 è inibito dal lattato in concentrazione superiori a 20-40 mM mentre M4 è inibito in misura minore da elevate concentrazioni di lattato.[20]
Distribuzione tissutale
Nell’uomo i differenti isoenzimi della lattato deidrogenasi hanno localizzazione tissutali preferenziali, che in genere riflettono il fenotipo metabolico del tessuto.
Infatti, tessuti con un fenotipo metabolico prevalentemente o esclusivamente aerobico, come il cuore, producono in misura maggiore isoenzimi a prevalenza della subunità H.[21]
Al contrario, nei tessuti dove il metabolismo anaerobico è importante, come fibre muscolari scheletriche nel corso di un esercizio inteso, o alcune zone scarsamente ossigenate nei tumori solidi, sono prodotti isoenzimi con prevalenza della subunità M.[5]
Esistono tuttavia eccezioni, come ad esempio il fegato, organo con fenotipo metabolico aerobico dove predomina LDH-5, ma dove l’ossidazione del lattato a piruvato, che può essere considerata come parte del ramo epatico del ciclo di Cori, è favorita dal basso rapporto NADH/NAD+ presente nel citosolico dell’epatocita.[13]
Di seguito una breve panoramica.
- LDH-1 o H4: muscolo cardiaco, rene e globuli rossi;
- LDH-2 o M1H3 ha una distribuzione simile a LDH-1;
- LDH-3 o M2H2: milza, cervello, globuli bianchi, rene e polmone;
- LDH-4 o M3H1: milza, polmone, muscolo scheletrico, globuli rossi e rene;
- LDH-5 o M4: fegato, muscolo scheletrico e polmone.
- LDH-6: sperma e testicolo
Nei singoli organi, LDH-1 e LDH-2 predominano nel cuore, nei reni e nei globuli rossi, LDH-3 nei polmoni, LDH-4 e LDH-5 nel muscolo scheletrico e LDH-5 nel fegato.
Gli isoenzimi presenti nel siero, e il cui dosaggio viene utilizzato a fini diagnostici, sono sempre di derivazione tissutale.[5]
Infine, in uno stesso tessuto/organo, i differenti isoenzimi possono essere presenti in quantità significative in differenti tipi cellulari, come ad esempio accade nel muscolo scheletrico, nel cervello, ma anche nei tumori solidi.
Ruolo
La lattato deidrogenasi è un enzima essenziale sia per la produzione che l’utilizzazione/rimozione del lattato.[4]
In condizioni ipossiche la cellula ricava l’ATP dall’ossidazione anaerobica del glucosio. A mezzo della via glicolitica il monosaccaride viene ossidato a due molecole di piruvato, ricavando da ciò 2 molecole di ATP e due di NADH. Tuttavia, affinché la glicolisi possa procedere, il NADH prodotto dovrà essere riossidato a NAD+, essendo il coenzima ossidato necessario alla reazione catalizzata dalla gliceraldeide-3-fosfato deidrogenasi (EC 1.2.1.12). Poiché in condizioni anaerobiche il complesso della piruvato deidrogenasi inibito e la fosforilazione ossidativa bloccata, l’ossidazione del NADH è portata a termine dalla reazione catalizzata dalla lattato deidrogenasi, che quindi consente alla glicolisi di procedere. Questa via metabolica porta quindi alla produzione di lattato dal glucosio, ed è nota come fermentazione lattica.[13][22]
L’acido lattico è spesso considerato un prodotto finale del metabolismo dei glucosio, e il suo accumulo nel corpo risulta essere pericoloso, potendo essere causa di acidosi lattica.[22] Ne consegue che la molecola deve essere rapidamente rimossa dai tessuti e dal circolo. Considerando ad esempio il lattosio prodotto dai globuli rossi, o quello prodotto dalle fibre muscolari che lavorano in condizioni di scarso apporto di ossigeno, come durante un esercizio intenso, questi può raggiungere, trasportato dal circolo ematico, tra gli altri, il fegato e il cuore. Nel fegato, sebbene LDH-5 favorisca la riduzione del lattato a piruvato, il basso rapporto NADH/NAD+ citosolico sposta l’equilibrio della reazione verso la formazione del piruvato, che potrà essere utilizzato per la produzione di energia, o entrare nella gluconeogenesi.[23] Nel cardiomiocita invece, l’isoenzima prevalente, LDH-1, ossida il lattato a piruvato che sarà utilizzato a fini energetici.
Bibliografia
- ^ a b c Berg J.M., Tymoczko J.L., and Stryer L. Biochemistry. 5th Edition. W. H. Freeman and Company, 2002
- ^ a b c Markert C.L., Shaklee J.B., Whitt G.S. Evolution of a gene. Multiple genes for LDH isozymes provide a model of the evolution of gene structure, function and regulation. Science 1975;189(4197):102-14. doi:10.1126/science.1138367
- ^ a b c Schueren F., Lingner T., George R., Hofhuis J., Dickel C., Gärtner J., Thoms S. Peroxisomal lactate dehydrogenase is generated by translational readthrough in mammals. Elife 2014;3:e03640. doi:10.7554/eLife.03640
- ^ a b c Laughton J.D., Charnay Y., Belloir B., Pellerin L., Magistretti P.J., Bouras C. Differential messenger RNA distribution of lactate dehydrogenase LDH-1 and LDH-5 isoforms in the rat brain. Neuroscience 2000;96(3):619-25. doi:10.1016/s0306-4522(99)00580-1
- ^ a b c d Farhana A., Lappin S.L. Biochemistry, lactate dehydrogenase. [Updated 2023 May 1]. In: StatPearls [Internet]. Treasure Island (FL): StatPearls Publishing; 2024 Jan-. Available from: https://www.ncbi.nlm.nih.gov/books/NBK557536/
- ^ Jin S., Chen X., Yang J., Ding J. Lactate dehydrogenase D is a general dehydrogenase for D-2-hydroxyacids and is associated with D-lactic acidosis. Nat Commun 2023;14(1):6638. doi:10.1038/s41467-023-42456-3
- ^ a b Krieg A.F., Rosenblum L.J., Henry J.B. Lactate dehydrogenase isozymes a comparison of pyruvate-to-lactate and lactate-to-pyruvate assays. Clin Chem 1967;13(3):196-203. doi:10.1093/clinchem/13.3.196
- ^ a b c Zinkham W.H., Blanco A., Clowry L.J. Jr. An unusual isozyme of lactic dehydrogenase in mature testes: localization, ontogeny, and kinetic properties. Ann N Y Acad Sci 1964;121:571-88. doi:10.1111/j.1749-6632.1964.tb14227.x
- ^ Palmer T., Bonner P.L.. Enzymes – Monomeric and oligomeric enzymes. 2nd Edition, Woodhead Publishing, 2011. doi:https://doi.org/10.1533/9780857099921.1.76
- ^ Bellamacina C.R. The nicotinamide dinucleotide binding motif: a comparison of nucleotide binding proteins. FASEB J 1996;10(11):1257-69. doi:10.1096/fasebj.10.11.8836039
- ^ a b Read J.A., Winter V.J., Eszes C.M., Sessions R.B., Brady R.L. Structural basis for altered activity of M- and H-isozyme forms of human lactate dehydrogenase. Proteins 2001;43(2):175-85.
- ^ Auerbach G., Ostendorp R., Prade L., Korndörfer I., Dams T., Huber R., Jaenicke R. Lactate dehydrogenase from the hyperthermophilic bacterium thermotoga maritima: the crystal structure at 2.1 A resolution reveals strategies for intrinsic protein stabilization. Structure 1998;6(6):769-81. doi:10.1016/s0969-2126(98)00078-1
- ^ a b c Forkasiewicz A., Dorociak M., Stach K., Szelachowski P., Tabola R., Augoff K. The usefulness of lactate dehydrogenase measurements in current oncological practice. Cell Mol Biol Lett 2020;25:35. doi:10.1186/s11658-020-00228-7
- ^ Moran L.A., Horton H.R., Scrimgeour K.G., Perry M.D. Principles of Biochemistry. 5th Edition. Pearson, 2012
- ^ Nelson D.L., M. M. Cox M.M. Lehninger. Principles of biochemistry. 6th Edition. W.H. Freeman and Company, 2012
- ^ Holmes R.S., Goldberg E. Computational analyses of mammalian lactate dehydrogenases: human, mouse, opossum and platypus LDHs. Comput Biol Chem 2009;33(5):379-85. doi:10.1016/j.compbiolchem.2009.07.006
- ^ Feng Y., Xiong Y., Qiao T., Li X., Jia L., Han Y. Lactate dehydrogenase A: a key player in carcinogenesis and potential target in cancer therapy. Cancer Med 2018;7(12):6124-6136. doi:10.1002/cam4.1820
- ^ Gray L.R., Tompkins S.C., Taylor E.B. Regulation of pyruvate metabolism and human disease. Cell Mol Life Sci 2014;71(14):2577-604. doi:10.1007/s00018-013-1539-2
- ^ Rogatzki M.J., Ferguson B.S., Goodwin M.L., Gladden L.B. Lactate is always the end product of glycolysis. Front Neurosci 2015;9:22. doi:10.3389/fnins.2015.00022
- ^ Stambaugh R., Post D. Substrate and product inhibition of rabbit muscle lactic dehydrogenase heart (H4) and muscle (M4) isozymes. J Biol Chem 1966;241(7):1462-7. doi:10.1016/S0021-9258(18)96733-5
- ^ Wroblewski F, Gregory KF. Lactic dehydrogenase isozymes and their distribution in normal tissues and plasma and in disease states. Ann N Y Acad Sci 1961;94:912-32. doi:10.1111/j.1749-6632.1961.tb35584.x
- ^ a b Li X., Yang Y., Zhang B., Lin X., Fu X., An Y., Zou Y., Wang J.X., Wang Z., Yu T. Lactate metabolism in human health and disease. Signal Transduct Target Ther 2022;7(1):305. doi:10.1038/s41392-022-01151-3
- ^ Gleeson T.T. Post-exercise lactate metabolism: a comparative review of sites, pathways, and regulation. Annu Rev Physiol 1996;58:565-81. doi:10.1146/annurev.ph.58.030196.003025