Le proteine, animali, vegetali, batteriche e virali, sono grandi polimeri lineari formati da centinaia e fino a migliaia di unità dette aminoacidi.
La sequenza con cui gli aminoacidi sono presenti, che è unica e determinata dal gene che codifica per la proteina in esame, è chiamata catena polipeptidica o struttura primaria. Nella catena polipeptidica gli aminoacidi sono legati in serie a mezzo di legami covalenti chiamati legami peptidici.
La struttura primaria determina in gran parte quella che sarà la struttura tridimensionale, o conformazione, della proteina.
Le proteine sono una classe di molecole molto grande, sono presenti in tutti gli esseri viventi, in tutti i compartimenti della cellula, e hanno strutture estremamente varie, anche all’interno di una stessa cellula, dove se ne possono ritrovare da centinaia a migliaia di tipi differenti, ognuno dei quali svolge una funzione specifica.
La grande varietà delle funzioni che sono in grado di svolgere deriva dalla possibilità della catena polipeptidica di ripiegarsi in strutture tridimensionali particolari che assicurano anche la capacità di legare molecole differenti. Si può quindi affermare che le proteine sono gli strumenti molecolari attraverso cui l’informazione genetica si esprime.
Indice
La scoperta
Grazie al fatto di essere più facili da isolare rispetto agli acidi nucleici, ai lipidi e ai polisaccaridi, lo studio delle proteine ha preceduto quello delle altre biomolecole, e può essere fatto risalire ai lavori sulla composizione chimica delle albumine condotti da Jöns Jacob Berzelius, uno dei padri della chimica moderna, e Gerardus Johannes Mulder, nel 1839. Per confronto, la scoperta del ruolo degli acidi nucleici nella trasmissione ed espressione dell’informazione genetica avvenne alla fine degli anni 40 del secolo scorso, quella del loro ruolo catalitico solo negli anni 80, mentre il ruolo dei lipidi nelle membrane biologiche venne alla luce negli anni 60.
Il termine proteina deriva dal greco proteios, che significa primario, e fu suggerito da Berzelius a Mulder. Berzelius riteneva infatti che le proteine potessero essere le sostanze biologiche più importanti.
Struttura
Al pari della altre macromolecole biologiche anche le proteine sono formate dall’unione di piccole molecole organiche, nel loro caso gli aminoacidi.
Sono stati individuati circa 20 aminoacidi differenti i quali, secondo la convenzione di Fischer, sono presenti quasi esclusivamente nella forma L. Occasionalmente, in proteine di origine batterica sono stati trovati D-aminoacidi.
Gli L-amminoacidi sono composti organici bifunzionali in quanto presentano un gruppo carbossilico e uno amminico legati a un atomo di carbonio, detto carbonio alfa, e per questo sono anche detti L-alfa-amminoacidi. Ogni amminoacido è identificato da un gruppo caratteristico, detto gruppo R, anch’esso legato al carbonio alfa. Il gruppo R è responsabile delle proprietà chimiche dell’amminoacido, in quanto possiede una varietà di dimensioni, carica, forme e reattività proprie.
Nel corso della sintesi della proteina il gruppo carbossilico di un amminoacido e quello amminico di un altro amminoacido sono legati covalentemente attraverso una reazione di condensazione catalizzata da specifici enzimi, ossia proteine dotate di attività catalitica. Nel corso della reazione si verifica il rilascio di una molecola d’acqua, con formazione di un legame peptidico. Si tratta di un legame rigido, planare e molto stabile in condizioni di pH fisiologico, tanto che in assenza di interventi esterni la sua vita è di circa 1100 anni. Dal legame end-to-end di molti amminoacidi si viene a formare lo scheletro polipeptidico della proteina.
Nella descrizione della struttura che assume nello spazio la catena polipeptidica è di aiuto distinguere vari livelli di organizzazione, ossia la struttura primaria, secondaria, le strutture supersecondarie, i domini, la struttura terziaria e quella quaternaria.
Nota: la convenzione di Fischer non è più utilizzata, se non per carboidrati e aminoacidi, sostituita dal sistema RS, il quale consente di assegnare in maniera più accurata il nome a molecole dotate di un centro di chiralità.
Struttura primaria
La prima proteina di cui è stata determinata la struttura primaria fu l’insulina, grazie al lavoro di Frederick Sanger.
La struttura primaria è la sequenza aminoacidica di una proteina, il suo livello di organizzazione più basso, e, come detto, è unica, geneticamente determinata, e a essa si deve la struttura tridimensionale e la funzione della proteina.
La struttura primaria può essere formata da 40 fino a oltre 4000 residui aminoacidici.
La catena polipeptidica è dotata di polarità in quanto le due estremità sono differenti: una presenta un gruppo amminico libero, detto gruppo ammino-terminale, l’altra un gruppo carbossilico libero, detto gruppo carbossi-terminale. Le due estremità della catena polipeptidica sono definite anche estremità N-terminale e C-terminale, per distinguere i due gruppi da quelli presenti nelle catene laterali degli aminoacidi interni alla catena. Per convenzione, l’estremità amminica libera è considerata l’inizio della catena, e si scrive a sinistra.
La struttura primaria è interessante anche perché, confrontando quella di una stessa proteina presente in specie differenti, si possono individuare le variazioni che il gene corrispondente ha subito, che sono un indice di quanto le specie si siano separate nel corso dell’evoluzione.
I termini dipeptide, tripeptide, oligopeptide e polipeptide sono utilizzati per indicare catene di lunghezza diversa, rispettivamente composte da 2, 3, meno di 50, e più di 50 residui.
Struttura secondaria
La scoperta della struttura secondaria delle proteine si deve al lavoro di Linus Pauling e Robert Corey, i quali proposero l’esistenza di due strutture dette alfa-elica e foglietto beta o struttura beta a foglietto pieghettato.
La struttura secondaria deriva dalla formazione di legami a idrogeno tra parti contigue della catena polipeptidica dotate di particolari sequenze aminoacidiche. Dunque, descrive la disposizione nello spazio di residui aminoacidici adiacenti lungo la struttura primaria.
Oltre alle strutture sopra citate ne sono state individuate altre quali i beta turn, i gamma turn e gli omega loop, tutte appartenenti al gruppo definito reverse turns. Queste strutture sono spesso presenti nei punti dove la catena polipeptidica inverte la propria direzione, e in genere sono disposte sulla superficie della molecola.
Circa il 32-38% degli aminoacidi delle proteine globulari si ritrova in una conformazione ad alfa-elica.
Le strutture successive a quella secondaria sono presenti solo nelle proteine globulari.
Strutture supersecondarie o motivi
Sono combinazione di strutture secondarie che formano una regione della molecola con una topologia e struttura tridimensionale caratteristica. Sono connesse tra di loro da regioni ad ansa prive di una struttura definita.
Tra i motivi più comuni si ritrovano l’alfa-alfa-corner, il beta-beta-corner, il beta-beta-hairpin, il beta-alfa-beta-motif, quest’ultimo spesso presenti nelle proteine che legano l’RNA o il DNA, e il 3beta-corner.
Domini
I domini sono regioni globulari che risultano dalla combinazione di motivi che si ripiegano in modo indipendente dal resto della catena polipeptidica a dare una struttura stabile.
Sono formati da 40-400 aminoacidi, tranne i domini motori e chinasici che presentano un numero molto maggiore di aminoacidi.
I domini sono stati classificati in tre gruppi principali, individuati in base alle strutture secondarie e motivi presenti:
- domini alfa;
- domini beta;
- domini alfa/beta.
Sono state identificate oltre 1000 famiglie di domini, e i membri di ciascuna famiglia sono detti omologhi.
Molto spesso un dominio è dotato di una specifica funzione, quindi è un’unità funzionale della proteina in cui è contenuto.
Le proteine possono essere costituite da un singolo dominio, quelle più piccole, o da più domini. Ad esempio, la chimotripsina (EC 3.4. 21.1), un enzima coinvolto nella digestione delle proteine, è composta da un dominio, mentre la papaina (EC 3.4.22.2) da due domini.
Struttura terziaria
La struttura terziaria, anche detta struttura nativa, è la struttura tridimensionale della proteina ed è la sua forma biologicamente attiva.
La prima proteina di cui è stata determinata tale struttura è stata la mioglobina, grazie al lavoro di John Kendrew.
La catena polipeptidica non è una struttura rigida ed è capace di ripiegarsi spontaneamente a dare una struttura tridimensionale, che in gran parte è determinata proprio dalla sua sequenza aminoacidica. Il ripiegamento della struttura primaria permette la transizione dal mondo unidimensionale della struttura primaria a quello tridimensionale della proteina.
In questo tipo di struttura, il ripiegamento della catena polipeptidica fa si che residui aminoacidici lontani si ritrovino vicini, dunque riguarda la disposizione nello spazio di aminoacidi lontani tra di loro nella struttura primaria.
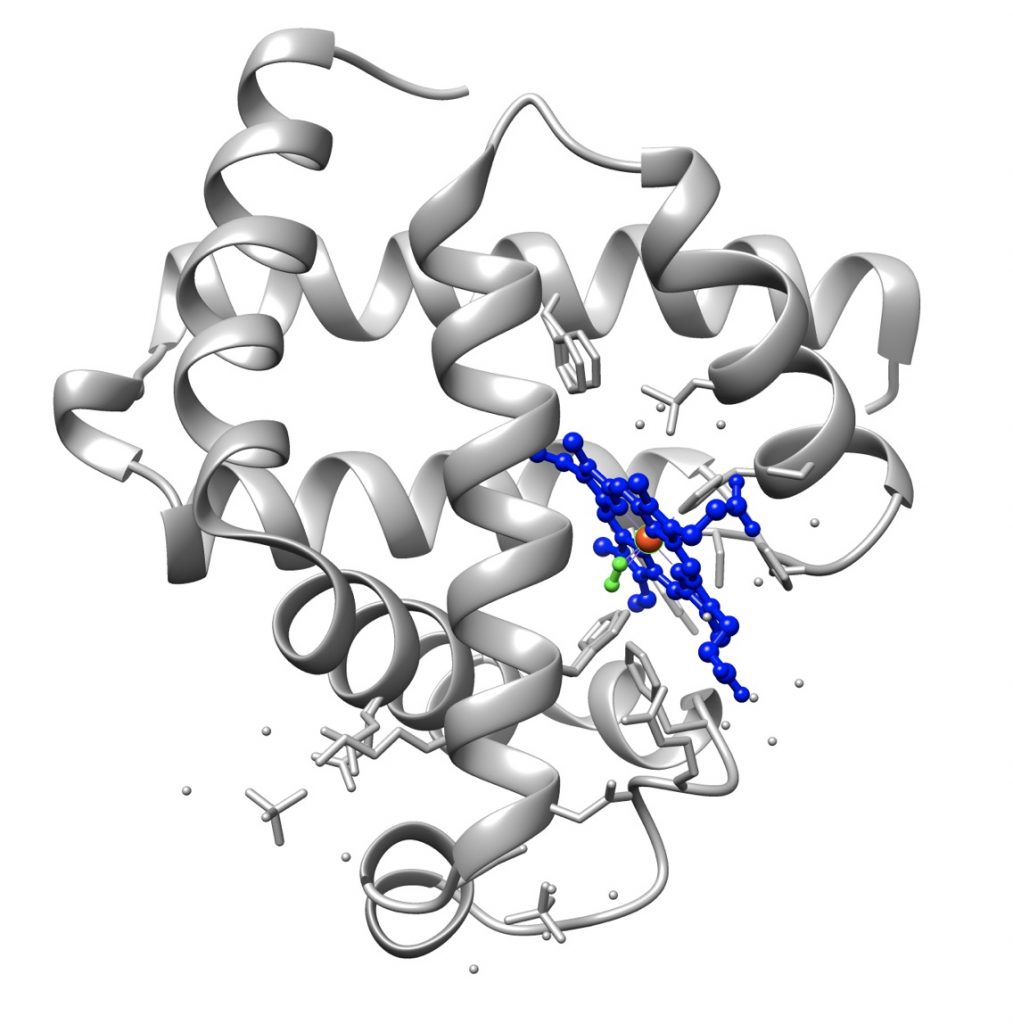
La struttura terziaria, specie delle proteine formate da più di 200 residui aminoacidici, è formata da diversi domini uniti da brevi segmenti polipeptidici, ed è spesso stabilizzata da legami disolfuro tra residui di cisteina, che si stabiliscono dopo che la molecola ha raggiunto la sua struttura nativa.
Non tutte le proteine globulari sono dotate di struttura terziaria. Un esempio sono le caseine del latte, la cui catena polipeptidica assume una conformazione tridimensionale disordinata, anche detta random coiled structure. La struttura tridimensionale disordinata fa si che queste proteine siano molto suscettibili all’attacco delle proteasi intestinali, e quindi alla liberazione degli aminoacidi costituenti, il che le rende estremamente idonee alla loro funzione nutrizionale. Un altro esempio di proteina random coiled è l’elastina, una delle più abbondanti proteine dell’organismo.
Struttura quaternaria
Questo ulteriore livello di organizzazione strutturale descrive come due o più catene polipeptidiche si associano a dare una singola struttura proteica. Dunque si riferisce alla disposizione spaziale delle singole catene e alla natura delle forze che le legano, quali:
- l’effetto idrofobo, che è anche il principale responsabile del ripiegamento delle proteine;
- i legami idrogeno;
- le interazioni di van der Waals;
- le interazioni ioniche;
- i legami covalenti trasversali.
La struttura risultante è definita oligomero o proteina oligomerica, e le subunità polipeptidiche costituenti, che possono essere uguali o diverse, monomeri o semplicemente subunità.
La maggior parte delle proteine intracellulari sono oligomeri, a differenza della maggior parte di quelle extracellulari. Un esempio di proteina con struttura quaternaria è l’emoglobina.
Questo livello di struttura è ovviamente assente nelle proteine globulari formate da una solo catena polipeptidica, ossia nelle proteine monomeriche.
Le proteine sono anche in grado di interagire tra di loro a formare strutture, dette macchine macromolecolari, nelle quali, agendo in modo sinergico, riescono a svolgere funzioni che da sole non sarebbero capaci di portare a termine. Esempi sono i complessi multienzimatici, come il complesso della piruvato deidrogenasi.
Funzioni
Le proteine sono le macromolecole più versatili presenti negli organismi viventi, nei quali hanno un ruolo centrale praticamente in tutte le strutture e funzioni cellulari, come:
- le reazioni chimiche;
- il trasporto di ossigeno;
- la risposta immunitaria;
- il controllo della crescita e della differenziazione;
- la trasmissione nervosa;
- il deposito;
- il supporto meccanico;
- il movimento.
Sono inoltre coinvolte nei processi digestivi che avvengono nel tratto gastrointestinale. Infatti, i macronutrienti, lipidi, carboidrati e le stesse proteine, assunti con l’alimentazione, per essere assorbiti devono essere idrolizzati rispettivamente ad acidi grassi, colesterolo, glicerolo, monosaccaridi e aminoacidi. Queste reazioni, nel corso della digestione dei carboidrati e delle proteine, come nella digestione dei lipidi, sono catalizzate da specifici enzimi, come l’alfa-amilasi (EC 3.2.1.1) per l’idrolisi dei legami glicosidici α-(1→4) di amilosio e amilopectina, i due polisaccaridi costituenti i granuli di amido.
Si noti che una delle metodiche per la classificazione delle proteine si basa proprio sulle loro funzioni biologiche.
Bibliografia
- Berg J.M., Tymoczko J.L., and Stryer L. Biochemistry. 5th Edition. W. H. Freeman and Company, 2002
- Fang C., Shang Y., Xu D. Improving protein gamma-turn prediction using inception capsule networks. Sci Rep 2018;8(1):15741. doi:10.1038/s41598-018-34114-2
- Garrett R.H., Grisham C.M. Biochemistry. 4th Edition. Brooks/Cole, Cengage Learning, 2010
- Lodish H., Berk A., Zipursky S.L., et al. Molecular cell biology. 4th edition. New York: W. H. Freeman; 2000. Section 3.1, Hierarchical Structure of Proteins.
- Kessel A., Ben-Tal N. Introduction to proteins: structure, function, and motion. CRC Press, 2011. doi:10.1002/cbic.201100254
- Milo R. What is the total number of protein molecules per cell volume? A call to rethink some published values. Bioessays 2013;35(12):1050-5. doi:10.1002/bies.201300066
- Moran L.A., Horton H.R., Scrimgeour K.G., Perry M.D. Principles of Biochemistry. 5th Edition. Pearson, 2012
- Nelson D.L., Cox M.M. Lehninger. Principles of biochemistry. 6th Edition. W.H. Freeman and Company, 2012
- Rudnev V.R., Kulikova L.I., Nikolsky K.S., Malsagova K.A., Kopylov A.T., Kaysheva A.L. Current approaches in supersecondary structures investigation. Int J Mol Sci 2021;22(21):11879. doi:10.3390/ijms222111879
- Stipanuk M.H., Caudill M.A. Biochemical, physiological, and molecular aspects of human nutrition. 3rd Edition. Elsevier health sciences, 2012
- Voet D. and Voet J.D. Biochemistry. 4th Edition. John Wiley J. & Sons, Inc. 2011